génétique

Italie: Découverte d'une tombe de bébé fille de 10000 ans
![]() Par
yvesh
Le 10/09/2022
Par
yvesh
Le 10/09/2022
Italie: Découverte d'une tombe de bébé fille de 10000 ans

Extrait du film en 3D de la sépulture découverte en Italie. https://doi.org/10.1038/s41598-021-02804-z
Une sépulture de bébé à Arma Veirana dans le nord-ouest de l'Italie donne un aperçu des pratiques funéraires et de la personnalité féminine au début de l'Europe mésolithique.
Résumé de la publication scientifique : L'évolution et le développement des comportements mortuaires humains revêtent une énorme importance culturelle. Nous rapportons ici une sépulture d'un jeune enfant richement décorée (AVH-1) à Arma Veirana (Ligurie, nord-ouest de l'Italie) qui est directement datée de entre 10 211–9 910 calibré BP (Avant le Présent, probabilité de 95,4 %), la plaçant au début de l'Holocène et donc attribuable au début du Mésolithique, une période culturelle à partir de laquelle les sépultures bien documentées sont extrêmement rares. L'histologie dentaire virtuelle, la protéomique et l'ADNa indiquent que le nourrisson était une fille âgée de 40 à 50 jours. Les artefacts associés indiquent un investissement matériel et émotionnel important dans l'inhumation de l'enfant. Le profil biologique détaillé d'AVH-1 établit l'enfant comme le premier quasi-nouveau-né européen documenté comme étant une femme. L'enterrement d'Arma Veirana donne ainsi un aperçu du statut social basé sur le sexe/genre.
Les pratiques mortuaires offrent une fenêtre sur les visions du monde et la structure sociale des sociétés passées. Ethnographiquement, de nombreuses cultures ont retardé l'attribution de la personnalité aux jeunes enfants, les maintenant dans un état liminal d'humanité 1 , 2 , 3 , 4 . Ainsi, le traitement funéraire des enfants fournit des informations importantes sur qui était considéré comme une personne et se voyait ainsi accorder les attributs d'un soi individuel, d'une agence morale et d'une éligibilité à l'appartenance à un groupe. En effet, d'importantes discussions persistent concernant la reconnaissance de la personnalité infantile chez les peuples préhistoriques 5 , 6 , 7. Nous rapportons ici l'inhumation d'un jeune enfant en Ligurie (nord-ouest de l'Italie) — Arma Veirana Hominin 1 (AVH-1 ; surnommée « Neve »), directement datée du début de l'Holocène. En Europe, le début de l'Holocène (à 11 700 cal BP) a largement coïncidé avec le début du Mésolithique, une période culturelle susceptible d'avoir catalysé d'importants changements sociaux alors que les humains s'adaptaient aux changements environnementaux importants après la fin de la dernière période glaciaire 8 , 9 . Les sépultures du début du Mésolithique sont extrêmement rares ou peu documentées 10 , 11 , 12 , 13 , 14, et AVH-1 apporte des données essentielles de cette période clé de la préhistoire. De manière significative, AVH-1 représente la première inhumation féminine proche du nouveau-né documentée en Eurasie et fournit de nouvelles informations sur la façon dont l'âge et le sexe/genre ont influencé la construction de la personnalité dans les sociétés préhistoriques de chasseurs-cueilleurs.
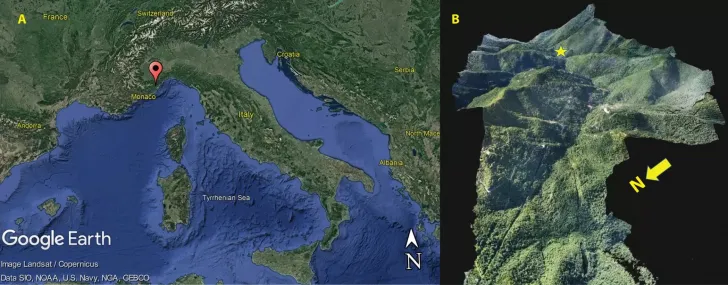
Fig 1 : Localisation et cadre du site d'Arma Veirana. ( A ) L'épingle rouge indique l'emplacement d'Arma Veirana situé dans le Val Neva (vallée de la rivière Neva) dans la région de la Ligurie (nord-ouest de l'Italie) - carte réalisée à l'aide de Google Earth Pro 7.3.4 ( https://earth.google. com ); ( B ) Modèle photogrammétrique 3D du Val Neva dans les Préalpes liguriennes généré à partir de photographies aériennes par le co-auteur DM (étoile = emplacement d'Arma Veirana). (Les auteurs et liens en bas d'article - cliquer pour agrandir l'image).
Arma Veirana est située dans les préalpes liguriennes (Fig. 1 ) à environ 15 km au nord-ouest de la ville d'Albenga (44°8′45.402″N, 8°4′18.85E). La grotte est située sur le côté nord de la vallée escarpée de Val Neva dans une falaise de marbre à une altitude de 451 m au-dessus du niveau de la mer. Arma Veirana conserve des gisements du Pléistocène supérieur (avec des horizons culturels Moustérien et Épigravettien supérieur) et du début de l'Holocène. Les informations supplémentaires comprennent des détails supplémentaires sur l'emplacement du site, la stratigraphie et la géoarchéologie, ainsi que des cartes du plan du site et l'historique des fouilles. En 2017 et 2018, des restes squelettiques in situ (tableau S8) et des artefacts associés ont été récupérés dans une fosse ovale de 15 cm de profondeur (< 800 cm2 dans la zone) taillée dans le dépôt sous-jacent de l'Épigravettien tardif (Figs. 2 , S9, S10). Le film S1 montre un survol d'un modèle photogrammétrique 3D de la grotte, y compris la position de l'excavation funéraire.
Lire la suite ci-dessous :

Indonésie: une nouvelle lignée humaine repérée
![]() Par
yvesh
Le 26/08/2021
Par
yvesh
Le 26/08/2021
Indonésie: une nouvelle lignée humaine repérée

La grotte de Leang Panninge sur la péninsule sud de Sulawesi, en Indonésie. Crédit : Projet de recherche Leang Panninge
Étude : Une femme indonésienne préhistorique appartenait à une lignée humaine éteinte
Enterrée dans une position fléchie avec de gros rochers, elle était génétiquement la plus proche des Australiens et des Papous indigènes - mais sa lignée était inconnue et semble avoir été perdue.
Une adolescente qui a vécu il y a 7 300 ans en Indonésie appartenait à une lignée inconnue d'humains modernes, qui s'est apparemment éteinte, a révélé mercredi 25 août 2021 une analyse publiée dans Nature.
L'histoire humaine est une histoire de migrations mystérieuses et de branches déconcertantes. Les ancêtres des Papous d'aujourd'hui et les ancêtres des Australiens indigènes se sont séparés il y a environ 37 000 ans. Cette fille trouvée dans la grotte de Leang Panninge à Sulawesi était, génétiquement, également liée aux deux, selon l'analyse.
En fait, son génome, le plus ancien d'un humain moderne de la région de Wallacea - les îles situées entre l'ouest de l'Indonésie et la Papouasie-Nouvelle-Guinée - indique une relation humaine ancienne jusqu'alors inconnue. Les chercheurs ont pu isoler suffisamment de matériel génétique du crâne d'un individu enterré il y a plus de 7 000 ans sur l'île indonésienne de Sulawesi. Il appartenait à une société de chasseurs-cueilleurs et a été enterré sur le site aujourd'hui appelé Leang Panninge ('Cave aux chauves-souris'). Une grande partie du code génétique correspondait à celui des Papouasie-Nouvelle-Guinée et des Aborigènes d'Australie d'aujourd'hui. Pourtant, des portions du génome ne correspondaient pas à ces groupes. Cela apporte de nouvelles surprises sur l'évolution de l'homme moderne.
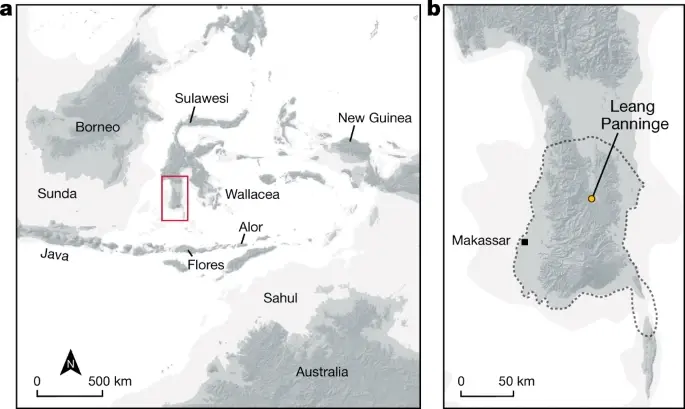
Fig. 1 : Emplacement du site d'étude. a , Sulawesi et Wallacea. Le rectangle rouge indique la région indiquée en b . b , Leang Panninge. La ligne pointillée indique la distribution du site toaléen.
L'étude internationale a été réalisée grâce à une étroite collaboration avec plusieurs chercheurs et institutions indonésiennes. Il était dirigé par le professeur Johannes Krause des instituts Max Planck d'anthropologie évolutive de Leipzig et de la science de l'histoire humaine à Iéna, le professeur Cosimo Posth du Senckenberg Center for Human Evolution and Palaeoenvironment de l'Université de Tübingen, et le professeur Adam Brumm de l'Université Griffith, Australie. L'étude a été publiée dans la dernière édition de Nature.
Les îles Wallacées ont formé des tremplins dans la propagation des premiers humains modernes de l'Eurasie à l'Océanie, il y a probablement plus de 50 000 ans. Les découvertes archéologiques montrent que les ancêtres de notre espèce vivaient à Wallacea il y a 47 000 ans. Pourtant, peu de squelettes humains ont été retrouvés. L'une des découvertes archéologiques les plus distinctives de cette région est le complexe technologique toaléen, daté d'une période beaucoup plus récente, il y a entre 8 000 et 1 500 ans. Parmi les objets fabriqués par les peuples de la culture toaléenne se trouvent les pointes de flèches en pierre caractéristiques connues sous le nom de pointes de Maros. La culture toaléenne n'a été trouvée que dans une zone relativement petite de la péninsule méridionale de Sulawesi. " Nous avons pu attribuer l'enterrement de Leang Panninge à cette culture ", explique Adam Brumm. "
Selina Carlhoff, candidate au doctorat à l'Institut Max Planck pour la science de l'histoire humaine et auteur principal de l'étude, a isolé l'ADN de l'os pétreux du crâne. " C'était un défi majeur, car les vestiges avaient été fortement dégradés par le climat tropical ", dit-elle. L'analyse a montré que l'individu de Leang Panninge était apparenté aux premiers humains modernes à se propager en Océanie depuis l'Eurasie il y a environ 50 000 ans. Comme le génome des habitants indigènes de Nouvelle-Guinée et d'Australie, le génome de l'individu de Leang Panninge contenait des traces d'ADN de Denisovan. Les Dénisoviens sont un groupe éteint d'humains archaïques connus principalement grâce à des découvertes en Sibérie et au Tibet. " Le fait que leurs gènes se trouvent chez les chasseurs-cueilleurs de Leang Panninge soutient notre hypothèse antérieure selon laquelle les Dénisoviens occupaient une bien plus grande zone géographique ", explique Johannes Krause.
Lire la suite ci-dessous:

Néandertalien: Art rupestre espagnol confirmé, groupes sanguins
![]() Par
yvesh
Le 03/08/2021
Par
yvesh
Le 03/08/2021
Néandertalien: Art rupestre espagnol confirmé
+ groupes sanguins examinés par la génétique

Cette combinaison d'images obtenues le 29 juillet 2021 montre une vue générale et un gros plan d'une tour de stalagmite partiellement colorée dans la grotte espagnole d'Ardales, dans le sud de l'Espagne.
Deux nouvelles études nous parlent des néandertaliens et confirment l'importance de notre cousin dans l'histoire humaine, d'autant plus qu'il est encore présent dans notre génome et y joue un rôle génétique pour une grande partie de l'humanité.
Une étude confirme que l'art rupestre espagnol ancien a été créé par les Néandertaliens
Les Néandertaliens, longtemps perçus comme étant simples et brutaux, ont vraiment peint des stalagmites dans une grotte espagnole il y a plus de 60 000 ans, selon une pré-publication du lundi 02 août 2021 d'une étude qui sera publiée dans les PNAS le 17 août 2021 : Le rôle symbolique du monde souterrain chez les Néandertaliens du Paléolithique moyen, PNAS (2021). DOI : 10.1073/pnas.2021495118
La question avait secoué la communauté paléoarchéologique depuis la publication d'un article de 2018 attribuant le pigment ocre rouge trouvé sur le dôme stalagmitique de la Cueva de Ardales à notre espèce "cousine" éteinte. La datation a suggéré que l'art avait au moins 64 800 ans, réalisé à une époque où les humains modernes n'habitaient pas le continent.
Mais la découverte était controversée, et "un article scientifique disait que ces pigments étaient peut-être une chose naturelle", un résultat du flux d'oxyde de fer, a déclaré à l'AFP Francesco d'Errico, co-auteur d'un nouvel article dans la revue PNAS.
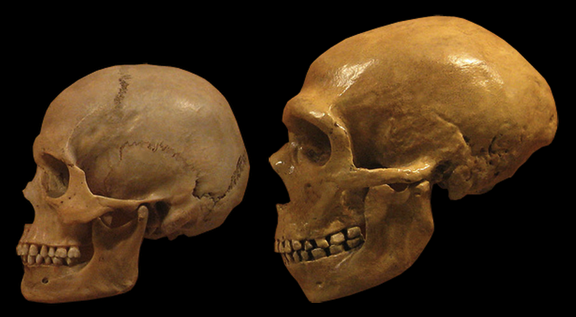
Comparaison entre un crâne d'Homo Sapiens (Homme Moderne à gauche) et un crâne de Homo Neanderthalensis (Homme de Néandertal à droite) - la moyenne du volume cervical des néandertaliens était plus élevée que la moyenne du volume cervical de l'homme moderne.
Cette nouvelle analyse a révélé que la composition et le placement des pigments n'étaient pas conformes aux processus naturels. Au lieu de cela, les pigments ont été appliqués par éclaboussures et soufflage. De plus, leur texture ne correspondait pas aux échantillons naturels prélevés dans les grottes, suggérant que les pigments provenaient d'une source externe.
Lire la suite ci-dessous :

Asie et Moyen-Orient: Deux nouvelles espèces humaines ?
![]() Par
yvesh
Le 26/06/2021
Par
yvesh
Le 26/06/2021
Asie et Moyen-Orient: Deux nouvelles espèces humaines ?

Vue d'artiste du Dragon Man dans son environnement. Crédit Chuang Zhao
Le même jour, deux publications, l'une chinoise et l'autre israélienne, revendiquent l'existence de deux nouvelles espèces apparentées au genre Homo (Nesher Ramla Homo et Homo longi). Et compte tenu des datations de ces fossiles, ces deux possibles nouveaux homininés auraient parfaitement pu rencontrer non seulement les néandertaliens mais aussi nos ascendants directs, Homo Sapiens sans parler de Dénisovien pour l'asiatique. Mieux, l'homininé asiatique serait même en fait notre plus proche cousin, raflant ce statut à néandertalien.
Pour rappel, certaines études génétiques ont affirmé qu'il manquait au moins l'ADN d'un homininé inconnu dans le génome de l'homme moderne, mais aussi des néandertaliens et denisoviens, il n'est donc pas impossible que l'un ou même les deux de ces nouvelles espèces fortement envisagées se soient aussi croisées avec nos propres ancêtres (l'être humain a en effet un certain pourcentage de son ADN actuel attribuable selon les régions à néandertalien et dénisovien:
Autre rappel, la génétique nous affirme depuis longtemps que Homo Sapiens (nous) et Néandertalien s'étaient tous deux séparés d'un unique ancêtre commun, avec une date qui recule au fil du temps (estimée à il y a environ 400 000 ans, puis 800 000 ans, l'estimation est maintenant de 1 million d'années.
Commençons par parler des études israéliennes, plus proche de l'Europe, qui sont toutes deux parues le 25 juin 2021 dans Science et qui s'appuient également sur d'autres publications :
Des fouilles dans un gouffre israélien ont dévoilé un groupe d'hominidés de l'âge de pierre jusqu'alors inconnu qui a contribué à l'évolution du genre humain (Homo). Les habitants d'un site appelé Nesher Ramla, qui vivaient il y a environ 140 000 à 120 000 ans, rejoignent les Néandertaliens et les Dénisoviens en tant que troisième population homo eurasienne qui s'est culturellement mêlée et peut-être croisée avec l'ancien Homo sapiens (amenant à l'homme moderne), selon les chercheurs, qui l'ont provisoirement nommé Nesher Ramla Homo.
De plus, les fossiles d'hominidés précédemment trouvés dans trois grottes israéliennes, qui datent d'il y a environ 420 000 ans, appartiennent probablement aussi à l'ancienne population représentée par les découvertes de Nesher Ramla, selon une équipe internationale dirigée par le paléoanthropologue Israel Hershkovitz.
« Nesher Ramla Homo était l'un des derniers survivants d'un ancien groupe [d'hominidés] qui a contribué à l'évolution des populations de Néandertaliens européens et d'Homo d'Asie de l'Est », explique Hershkovitz. (YH : cette nouvelle espèce, pour l'instant, aurait donc vécu de 420 000 ans à 120 000 ans environ, localement).
Les travaux de Nesher Ramla ont mis au jour cinq morceaux d'une boîte crânienne et une mâchoire inférieure presque complète contenant une dent molaire. Ces fossiles ressemblent à certains égards aux Néandertaliens et à d'autres hominidés rappellant certains fossiles souvent classés à tord ou à raison comme Homo heidelbergensis, une espèce pré-néandertalienne qui aurait occupé des parties de l'Afrique, de l'Europe et peut-être de l'Asie de l'Est depuis environ 700 000 ans ( SN : 15/5/19 ).
Des parties d'une mâchoire (à gauche) et d'une boîte crânienne (à droite), trouvées sur le site israélien de Nesher Ramla, représentent une ancienne population d'hominidés qui a contribué à l'évolution des Néandertaliens européens et peut-être d'anciens groupes Homo en Asie de l'Est, selon les chercheurs. AVI LEVIN ET ILAN THEILER/FACULTÉ DE MÉDECINE SACKLER/TEL AVIV UNIV.
Dans les sédiments fossilifères, l'équipe de Hershkovitz a déterré environ 6 000 artefacts en pierre et plusieurs milliers d'os de gazelles, de chevaux, de tortues et d'autres animaux. Certains de ces os contenaient des marques d'outils en pierre faites lors de l'enlèvement de la viande.
Des combinaisons de traits sur certains fossiles d'Hominidés chinois, y compris une mâchoire d'enfant datant peut-être de plus de 200 000 ans (YH : et ayant une croissance des dents identique à celle de l'homme moderne !), ressemblent à l'apparence des nouveaux fossiles israéliens, dit Hershkovitz ( SN: 1/16/19 ). Les anciens groupes Homo ayant des racines à Nesher Ramla ont peut-être atteint l'Asie de l'Est et se sont peut-être mariés avec certains groupes qui y vivent déjà, spécule-t-il.
Mais Nesher Ramla Homo n'a pas eu à aller jusqu'en Asie de l'Est pour interagir avec d'autres groupes d'hominidés. Les outils en pierre trouvés avec les fossiles de Nesher Ramla Homo correspondent à des outils d'âge comparable fabriqués à partir de morceaux de roche préparés par H. sapiens à proximité ( SN: 1/25/18). Nesher Ramla Homo et H. sapiens doivent avoir échangé des connaissances sur la fabrication d'outils en pierre, et peut-être se sont mélangés, dit Hershkovitz. Les tentatives d'extraction de l'ADN des fossiles de Nesher Ramla, qui révéleraient si des croisements ont eu lieu, ont échoué.
Il est intrigant que des outils en pierre généralement associés à H. sapiens aient été trouvés avec des fossiles aussi distinctifs, explique le paléoanthropologue John Hawks de l'Université du Wisconsin-Madison, qui n'a pas participé à la nouvelle recherche. " Ce n'est pas un flingue qui prouve qu'il y a eu des interactions étroites entre Nesher Ramla Homo et Homo sapiens, mais c'est très suggestif." YH : et il est bien sûr impossible de connaître les contributions de Néandertalien à l'Homo Sapiens et inversement (ainsi que celles de Denisovien et autres d'ailleurs)

Les outils en pierre fabriqués par une ancienne population Homo au Moyen-Orient (illustrés) ressemblent à ceux fabriqués à la même époque par les Homo sapiens voisins, suggérant que les deux groupes avaient des contacts étroits.TAL ROGOVSKI
Les preuves de Nesher Ramla correspondent à un scénario dans lequel le genre Homo a évolué en tant que populations et espèces étroitement liées du Pléistocène moyen, y compris les Néandertaliens, les Denisoviens et H. sapiens. Des groupes basés dans des régions méridionales habitables se sont déplacés dans une grande partie de l'Europe et de l'Asie pendant des étendues relativement chaudes et humides, écrit la paléoanthropologue Marta Mirazón Lahr de l'Université de Cambridge dans un commentaire publié avec les nouvelles études. Ces anciens groupes se sont croisés, se sont fragmentés, se sont éteints ou se sont recombinés avec d'autres groupes Homo en cours de route, produisant une variété d'apparences squelettiques observées dans les fossiles d' Homo européens et est-asiatiques, suggère Lahr.
Le mélange génétique et culturel des groupes d'Homo eurasiens au cours de la période du Pléistocène moyen – qui s'étendait d'environ 789 000 à 130 000 ans – s'est produit trop fréquemment pour permettre l'évolution d'une espèce distincte dans ce cas, selon l'équipe.
Ces fossiles compliquent encore l'arbre généalogique humain, qui est devenu plus complexe ces dernières années avec des ajouts tels que H. naledi d'Afrique du Sud et le H. luzonensis proposé aux Philippines ( SN : 9/10/15 ; SN : 4/10 /19 ). YH : Sans oublier que plusieurs autres fossiles, comme cette machoire pêchée du côté de Taïwan en 2015 ou d'autres en Asie, posent également le même type de question.
« C’est un hominidé plus archaïque que Néandertal, mais c’est le premier hominidé découvert qui utilise un type d’outil de pierre utilisé par Sapiens », explique l’une des coauteures de l’étude publiée jeudi dans la revue Science, Marion Prévost, de l’Université hébraïque de Jérusalem. Cela ouvre la porte à la possibilité que les néandertaliens, qui ont vécu en Europe jusqu’à il y a 40 000 ans (YH : Non, tout indique qu'on trouve encore néandertalien il y a 29 000 ans et peut-être moins dans le sud de l'Europe), aient appris d’autres techniques de taille de la pierre de l’homme de Nesher Ramla plutôt que de Sapiens. « Il est possible que Nesher Ramla ait migré vers l’Europe et engendré le néandertalien », dit Mme Prévost.
Le type d’outil jusqu’à maintenant propre à Sapiens, qui a aussi été découvert sur le site israélien, s’appelle une coupe de pierre « levallois centripète ».
Un autre outil trouvé sur le site a attiré l’attention des paléontologues. « C’est un outil qu’on ne retrouve nulle part ailleurs au Levant, qui a deux types de coupes le long du manche, pour servir à la fois de couteau et de racloir, dit Mme Prévost. On retrouve ce type d’outil ailleurs dans le monde. Il s’agit probablement d’une évolution technologique indépendante. » Ce type d’outil est plus ancien que ceux qu’utilisaient Homo sapiens.
Le site de Nesher Ramla - PHOTO FOURNIE PAR YOSSI ZAIDNER
Le site a été découvert en 2010 lors du creusage d’une carrière, et des fouilles de sauvetage ont été faites jusqu’en 2012. Les paléontologues font leurs analyses depuis. « Nous avons 80 000 outils de pierre à examiner, alors nous n’avons pas fini », dit Mme Prévost. L’homme de Nesher Ramla présente quelques similitudes avec un autre hominidé archaïque retrouvé en Israël dans la caverne de Qesem, découverte en 2000, qui n’a jamais été formellement identifié. Il pourrait aussi présenter des similitudes avec des individus d’autres sites mis au jour dans la péninsule arabique, selon Mme Prévost.
Voir les sources en bas de l'article.
Lire la suite ci-dessous pour la nouvelle espèce revendiquée en Chine :

Une vaste étude confirme la singularité génétique basque
![]() Par
yvesh
Le 10/06/2021
Par
yvesh
Le 10/06/2021
Une vaste étude confirme la singularité génétique basque

Crédit André Flores-Bello (EL PAÍS) Représentation de la structure génétique dans le territoire basque historique, où le vert représente l'ascendance basque, et le bleu et le rouge, le mélange avec les communautés voisines.
La plus grande étude jamais réalisée sur près de 2 000 échantillons d'ADN réalisée par des chercheurs de l'université Pompeu Fabra (UPF) de Barcelone a confirmé la "singularité génétique" des Basques en Europe. L'enquête, cependant, a révélé que cette différence n'a commencé à émerger qu'il y a 2 500 ans, à l'âge du fer. " Nos analyses confirment que les Basques ont été influencés par les grandes vagues migratoires en Europe jusqu'à l'âge du fer, selon un schéma similaire à celui de leurs populations environnantes ", expliquent les auteurs dans l'étude publiée dans la revue Current Biology.
Disons tout de suite que l'histoire de la génétique basque est jonchée de résultats contradictoires. L'un des co-auteurs de la nouvelle étude, Jaume Bertranpetit, avait déjà mené des recherches en 2010 qui étaient parvenues à la conclusion inverse : que les Basques ne présentaient pas une unicité génétique. " Sans aucun doute, nos précédents travaux n'étaient pas corrects ", reconnaît Bertranpetit. Son groupe avait utilisé des techniques d'analyse génétique qui ont maintenant été dépassées par les nouvelles technologies (YH : qui seront à leur tour dépassées tôt ou tard).
L' origine des Basques fascine la communauté scientifique depuis le XIXe siècle. L'anthropologue français Paul Broca s'est faufilé dans un cimetière basque une nuit de 1862 pour voler des crânes qu'il voulait étudier pour leurs supposées différences génétiques. Juan José Ibarretxe, premier ministre du gouvernement régional basque jusqu'en 2009, a proclamé que le peuple basque « existe depuis 7 000 ans » pour promouvoir sa vision d'un État basque indépendant. Et le président de l'époque du Parti nationaliste basque (PNV), Xabier Arzalluz, a affirmé en 2000 que les Basques étaient « les plus anciens habitants d'Europe », avec « leurs propres racines » depuis la préhistoire.
Bien que les différences génétiques basques soient notables, l'étude montre que c'est le résultat de siècles d'isolement et de consanguinité potentiellement causés par des dialectes basques uniques qui n'ont aucun croisement avec d'autres langues européennes, ni même avec aucune autre langue vivante aujourd'hui. YH : notons aussi tout de suite que plusieurs linguistes ne semblent pas d'accord avec ce postulat, ni même d'ailleurs d'accord avec les origines et datations de certaines migrations, voir ci-dessous.
Leur langue limitait leur interaction avec les autres communautés , qui ne pouvaient pas les comprendre. " Nos analyses soutiennent l'idée que l'unicité génétique des Basques ne peut pas être attribuée à une origine différente par rapport aux autres populations ibériques, mais plutôt à un flux de gènes externes réduit et irrégulier depuis l'âge du fer ", indique l'étude. Cela signifie que « les Basques ne sont pas des Martiens », plaisante David Comas, auteur principal de l'étude et professeur d'anthropologie biologique au Département des sciences expérimentales et de la santé (DCEXS) de l'UPF.
Il y a environ 7 000 ans, des groupes génétiquement très proches des agriculteurs et éleveurs néolithiques d'Anatolie sont arrivés dans la péninsule ibérique. Leur mélange avec les populations indigènes a laissé des groupes dont l'ascendance était à 80 % néolithique et 20 % mésolithique local, selon le généticien basque Íñigo Olalde, dont l'équipe de l'université Harvard a reconstitué l' histoire génomique de la péninsule ibérique en 2019.
Par la suite, il y a environ 4 500 ans, les nomades qui ont quitté les steppes de la Russie actuelle ont commencé à arriver dans la péninsule ibérique, remixant les gènes de la population dans un équilibre de 40% d'ascendance étrangère et 60% de ce qui s'y trouvait déjà. C'est le substrat génétique commun à tous les peuples de la péninsule ibérique, y compris les Basques.
Cette étude récente, alliant la linguistique et la génétique ne semble pas dire la même chose au niveau des datations et origines réelles de certaines migrations en Europe de l'ouest et y est aussi cité d'autres études, comme celle-ci: "Une étude canadienne, The Homo Neanderthalis and the Dravidians : A Common Origin and Relation to Harappan Civilisation and Vedas, estime que les Dravidiens, Sumériens, Egyptiens, Etrusques, Celtes et Basques avaient une même origine, avaient conservé des gènes néanderthaliens - en particulier les Basques - présentaient une même déficience du métabolisme du cholestérol à l’origine d’autres déficiences génétiques, parlaient et écrivaient une langue commune akkado-dravidienne et avaient adopté un modèle semblable de société matriarcale basé sur le culte de la Déesse-mère." :
La carte ci-dessus, réalisée dans le cadre d’un projet géno-géographique de National Geographic financé par IBM, résume bien ces migrations et le rôle majeur que l’Inde a joué dans la diffusion d’une civilisation venue d’Afrique de l’Est tant vers l’Asie du Sud-Est que vers les steppes d’Asie centrale, l’Europe et l’Afrique du Nord. (Source : Genographic Project web site. http://www-03.ibm.com/press/us/en/photo/35881.wss )
Alors que d'autres linguistes affirment que le Basque est considéré comme l'une des 4 langues en provenance directe de... l'Oural, dans le Caucase Russe. Ce qui d'ailleurs n'est pas gênant en soit puisque l'Oural est aussi considéré comme étant un lieu de passage des mêmes migrations provenant d'Asie Centrale, avec donc un endroit où les gênes et les langues se sont mélangés...
Olalde affirme que les Basques sont génétiquement différents du reste de la péninsule ibérique – mais pas si différents. « Il y a des décennies, on disait que les Basques étaient la continuation des chasseurs-cueilleurs, puis on a vu que c'était un mensonge total. Ensuite, on a dit qu'ils étaient une continuation du peuple néolithique qui est venu plus tard et cela s'est également avéré faux », explique-t-il.
Lire la suite ci-dessous :

Angleterre: Les bateaux agés de 4000 ans de Ferriby
![]() Par
yvesh
Le 03/06/2021
Par
yvesh
Le 03/06/2021
Angleterre: Les bateaux agés de 4000 ans de Ferriby
https://www.penn.museum/sites/expedition/the-bronze-age-boats-of-north-ferriby-yorkshire/
Retour donc sur la navigation préhistorique et protohistorique, pour accompagner le changement de consensus en cours au sujet de l'ancienneté de la navigation (humaine et préhumaine) grâce aux découvertes et aux études scientifiques liées. Je vais parler donc aujourd'hui des bateaux de Ferriby, dont les dernières datations (calibrées C14 en 2001) font remonter le plus ancien à plus de 2000 ans avant JC. Et on ne parle pas ici de pirogues préhistoriques mais bien de bateaux en bois cousus.
Les plus anciens bateaux d'Europe construits en planches de l'âge du bronze ont été trouvés près des estuaires britanniques et des voies maritimes. Ces lieux de découverte montrent qu'ils faisaient du commerce et échangeaient des marchandises et pouvaient également prendre la mer. L'ambre et le métal étaient échangés depuis la mer du Nord et le canal (la Manche) où les marchandises de prestige allaient, les idées suivaient. Les constructeurs de bateaux étaient des techniciens, des marins et ils étaient des pionniers, des explorateurs. Autant le dire tout de suite : les navires cousus identiques semblent avoir existé aussi en Méditerranée, faisant de cette technique une sorte d'héritage mondial, mais que le plus ancien découvert est plus récent que ceux d'Angleterre. Les plus anciens étant probablement les asiatiques (inuits et sibériens) et aussi probablement à l'origine de l'héritage mondial concernant la navigation de l'homme moderne, via le nord de l'Europe notamment. A priori, le plus ancien bateau cousu de Méditerranée, découvert à Zambratija en 2014, date du 7e siècle av. J.-C seulement. On constate aussi que les techniques sont inchangées pendant de nombreux siècles à leur sujet, alors que d'autres techniques apparaissent en parallèle.
D'une façon certaine et archéologique, les premiers bateaux de l'homme moderne datent du néolithique, des pirogues monoxyles, mais d'autres types de bateaux (de type skiff) ont été conçus également. Il semblerait aussi que des radeaux s'appuyant sur des outres ou des amphores, inspirés pour le transport de ces dernières ou non, émergent au Moyen-Orient : en Mésopotamie, en Égypte ou en Crète selon l'interprétation que l'on fait de certaines gravures. On a par exemple retrouvé à Noyen-sur-Seine en France une pirogue monoxyle, fabriquée dans un tronc de pin sylvestre et conservée sur une longueur de plus de 4 mètres, datée du mésolithique (-7000 av. J.-C.), et actuellement visible au musée de Nemours. Deux autres pirogues, disposées côte-à-côte et en excellent état, ont également été découvertes en amont de Paris, à Nandy en France, et sont datées de 7245-6710 av. J.-C. pour la plus ancienne, 7040-6620 av. J.-C. pour l'autre, ce qui en ferait a priori les plus vieux bateaux découverts sur le plan mondial. Archives: Un bateau en roseau vieux de 7 000 ans a été découvert au Koweït. En fait, le plus ancien bateau jamais découvert, le canoë Pesse, a été trouvé aux Pays-Bas, c’est une pirogue faite à partir de tronc d’arbre évidé de Pin sylvestre (Pinus sylvestris) qui a été construit entre 8200 et 7600 av. J.-C. (entre 10200 et 9600 ans avant maintenant donc) Ce canoë est exposé au Drents Museum à Assen, aux Pays-Bas. Notons que la découverte récente d'un chantier naval daté du mésolithique, en Angleterre également, apporte également une grande pièce archéologique à la navigation préhistorique précoce, y compris maritime.
Les bateaux de Ferriby sont datés eux du début de l'âge du bronze et ont été découverts en 1937 sur la rive nord de l'Humber, à proximité de North Ferriby dans l'East Yorkshire en Angleterre. Des planches furent remarquées dans l'estran de l'Humber en 1937 par Edward et Ted Wright, des fouilles eurent lieu à partir de 1938 effectuées par Philip Corder, mettant au jour entre 1939 et 1940 les vestiges du premier bateau, Ferriby-1. En novembre 1940 fut mise au jour la planche-quille du deuxième bateau, Ferriby-2, par les frères Wright, jusqu'à ce que la guerre oblige l'arrêt des fouilles.

Edward Wright, Les Ferriby Boats: Seacraft of the Bronze Age. 1990, Routledge, Londres.
À la fin de la guerre, en 1946, les fouilles sont reprises et les restes des barques (Ferriby-1 et Ferriby-2) sont alors découpés pour être amenés au musée national de la Marine à Greenwich, où elles sont datées de l'Âge du Bronze. En 1963, Ted Wright découvre les restes d'un troisième bateau, Ferriby-3, qui sont retirés et amenés au musée de Hull. En 1984 et 1989, sont découverts quelques rares restes de deux autres bateaux, Ferriby-4 et Ferriby-5.
Lors d'une nouvelle datation effectuée en mars 2001, ces restes furent alors proclamés comme étant les plus anciennes barques assemblées d'Europe.
Lire la suite ci-dessous :